在肿瘤学的研究中,致癌基因如何影响信号通路变化是人们对致癌基因造成肿瘤发生的机制认知的关键所在,早期通过应用抗体或芯片技术的靶向蛋白研究受限于分析广度与深度,无法解析更全面的蛋白调控变化,也无法揭示蛋白质翻译后修饰在其中的角色功能。因此,利用基于质谱的高深度蛋白质组学以及蛋白质修饰组学并整合转录组等测序技术的多维组学分析,提供了全景式解析肿瘤发生过程中蛋白调控网络变化的热门系统生物学手段。
2019年8月16日,来自美国孟菲斯St. Jude Children' s Research Hospital的研究员在国际知名期刊Nature Communications上发表了多组学驱动的肿瘤驱动基因信号通路调控的文章,研究者以HGG小鼠模型为研究对象,采用高深度蛋白质组学、磷酸化修饰组学和转录组学的多组学整合研究,比较了两种由致癌受体酪氨酸激酶(RTKs:PDGFRA D842V和TPM3-NTRK1融合)驱动的HGG小鼠模型。本项研究工作进一步证实了以蛋白质组学为核心的多组学整合分析在肿瘤等复杂疾病成因分析中的优势,并提供了新的肿瘤潜在治疗思路。

研究者首先对两种不同的HGG模型小鼠的肿瘤样本进行多维组学分析,其中应用TMT标记定量的高深度蛋白质组学与磷酸化修饰组学分别鉴定到13860种蛋白以及30431个磷酸化修饰位点。这组数据达到了非常高的鉴定深度,为后续多维组学的关联分析提供了保障。
随后,研究者分别在蛋白质组学和磷酸化修饰组学以及转录组三个层次的数据水平上分析两种HGG肿瘤模型以及对照组的数据分离度,通过PCA分析以及表达聚类分析,发现三个不同组别的样本在RNA、蛋白以及磷酸化层面均具有明显的分离。同时研究者也再次通过蛋白组与转录组的关联分析证实了二者之间关联性一般。
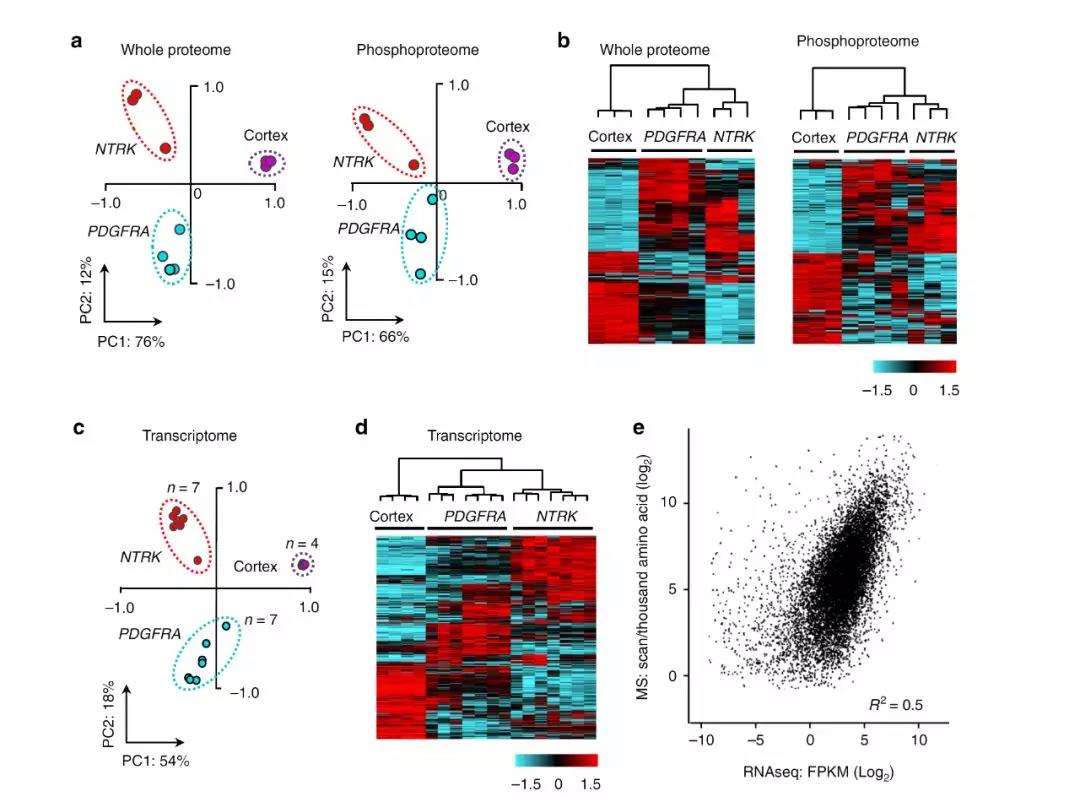
图2 不同样本在多维组学层面的分离聚类
接下来,研究者分别在三个不同组学的维度进行差异表达分析,通过WGCNA以及one-way ANOVA等分析技术,研究者找到了不同组学维度上两种HGG肿瘤样本中的特征调控通路。考虑到信号调控通路中磷酸化与激酶之间的相互作用,研究者进一步分析了不同肿瘤样本上磷酸激酶的活性分析。研究者发现,在HGG模型中激酶的活性相较于对照组有着较大的不同,证明了肿瘤中激酶表达谱以及活性的重编程,其中最值得关注的是AKT激酶以及其下游磷酸化信号通路具有较强的激活。
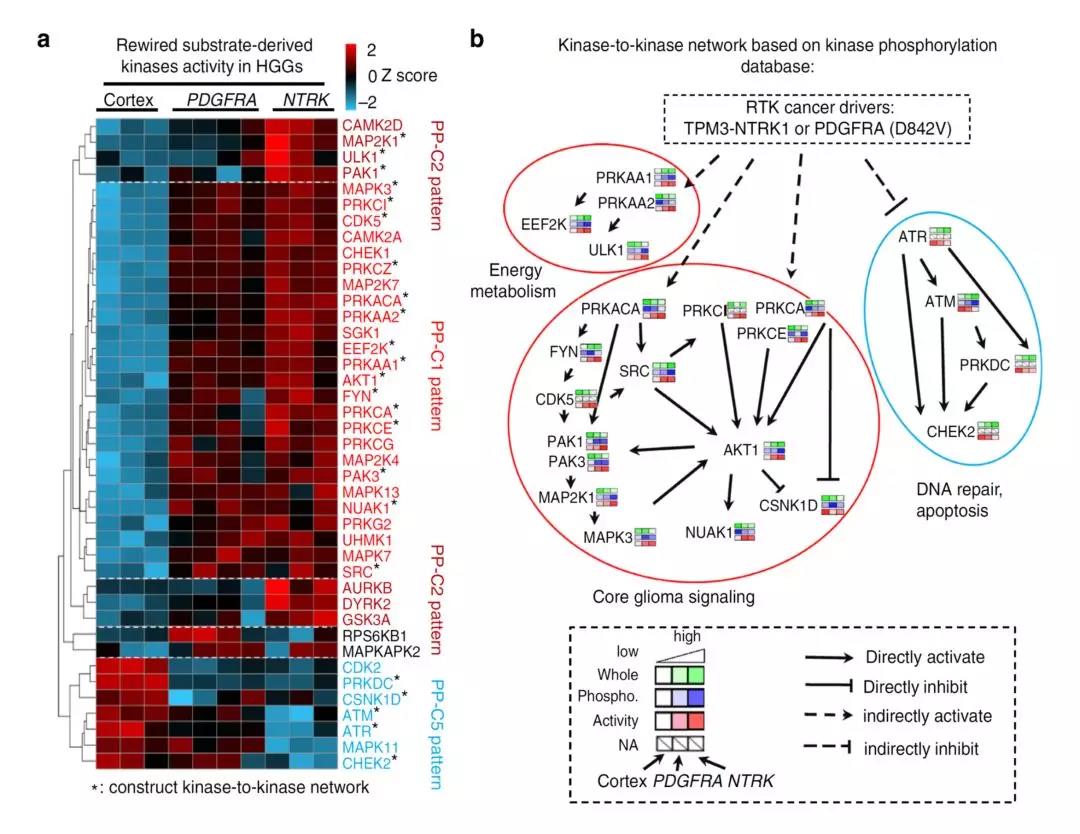
图3 激酶活性分析证实了HGG肿瘤模型中揭示了AKT激酶以及其下游信号通路的关键性
随后研究者进一步分析了转录组数据中与激酶相关的转录因子表达水平变化,得到HGG肿瘤中特异性的激酶-转录因子调控网络,并且发现相较于PDGFRA突变,NTRK突变的HGG肿瘤模型表现出更强的AKT激活活性,且具有更差的预后。
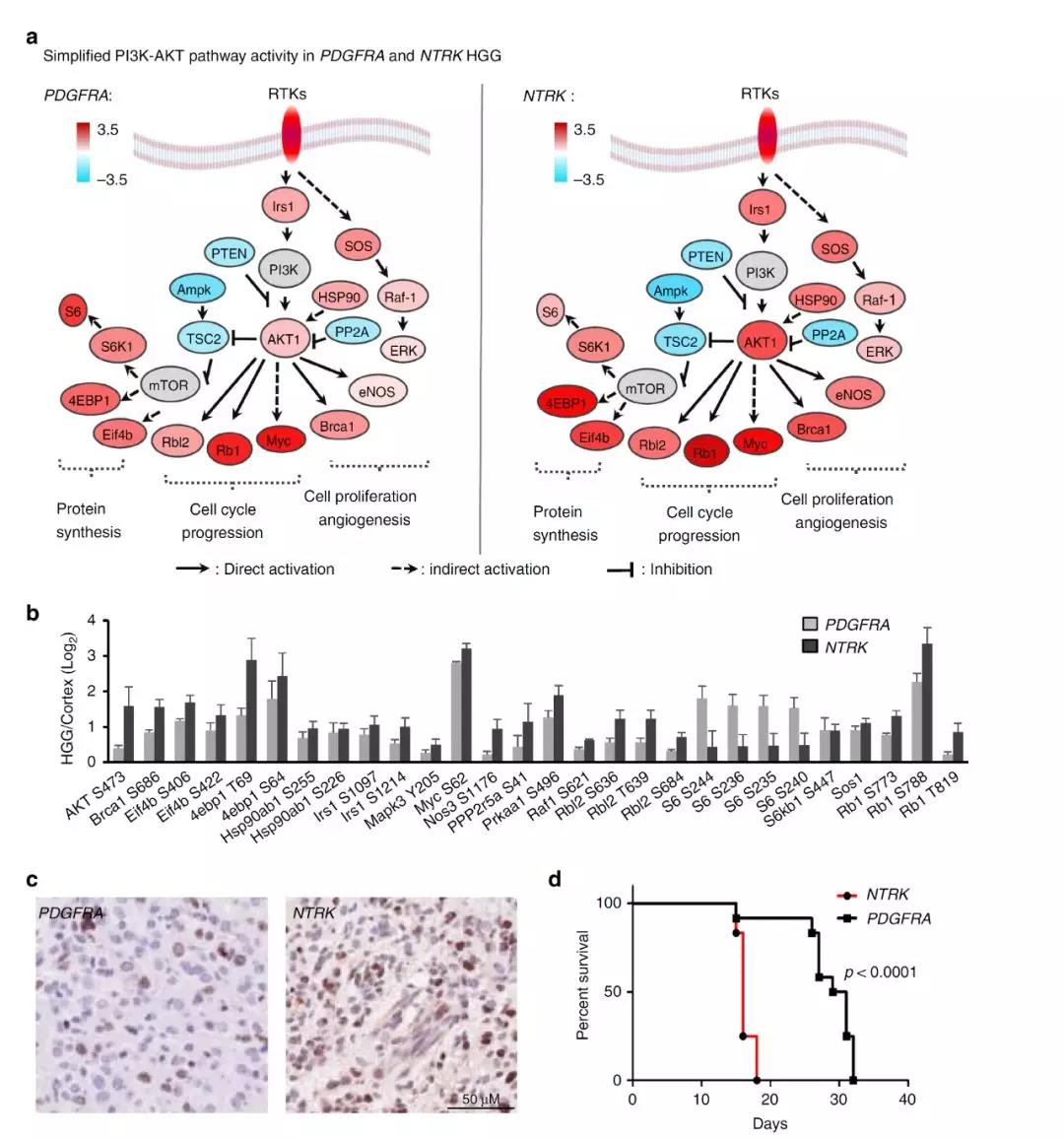
图4 NTRK与PDGFRA突变的HGG模型差异
最后,研究者结合上述多维组学的数据,通过CRISPR-Cas9的方法对发现的一些关键作用基因如转录因子或关键激酶等分别进行敲除筛选,寻找到潜在的肿瘤治疗靶点,为后续转化医学研究提供新思路。
本篇多维组学大数据文章充分展示了利用强有力的系统生物学分析手段并结合高质量的深度覆盖蛋白质组学、修饰组学以及转录组学数据能够全景式呈现蛋白调控网络变化的全貌,进而发现新的调控通路或调控模式,使我们的研究不再是管中窥豹,而是纵览全局,一目了然。
参考文献:Wang H, et al., 2019, Deep multiomics profiling of brain tumors identifies signaling networks downstream of cancer driver genes. Nat Commun.
转载于 Dr.Proteomics 精准医学与蛋白组学





 浙公网安备33010802007965号
浙公网安备33010802007965号